Představujeme produkt SALSA® MLPA® probemix P093-C2 HHT/HPAH

Seznamte se s novou verzí produktu SALSA® MLPA® probemix P093-C2 HHT/HPAH C2. V porovnání s verzí C1 byla upravena délka několika sond.
Katalogová čísla:
- P093-025R: SALSA® MLPA® probemix P093 HHT/HPAH, 25 reakcí.
- P093-050R: SALSA® MLPA® probemix P093 HHT/HPAH, 50 reakcí.
- P093-100R: SALSA® MLPA® probemix P093 HHT/HPAH, 100 reakcí.
K použití v kombinaci s reagenčním SALSA® MLPA® kitem, dostupný pro různý počet reakcí. MLPA reagenční kity jsou k dispozici s FAM nebo Cy5.0 značenými primery, vhodné pro kapilární sekvenátory Applied Biosystems a Beckman.
Certifikát analýzy: Informace o podmínkách skladování, testech kvality a vzoru elektroforeogramu z aktuální šarže je k dispozici na adrese mlpa.com.
Bezpečnostní opatření a varování: Pouze pro profesionální použití. Vždy si před použitím pročtěte nejnovější popis produktu a MLPA obecný protokol. Je odpovědností uživatele, aby se seznámil s nejnovějšími vědeckými poznatky, než bude vyvozovat závěry z výsledků získaných s tímto produktem.
Účel použití SALSA® MLPA® probemix P093-C2 HHT/HPAH
Salsa MLPA kit P093 HHT/HPAH je test pro in vitro diagnostiku (IVD)1 nebo pouze pro výzkumné účely (RUO) a slouží pro detekci delecí nebo duplikací ENG, ACVRL1 a BMPR2 s cílem potvrdit klinickou diagnózu hereditární hemoragické teleangiektázie (HHT) nebo dědičné plicní arteriální hypertenze (HPAH), nebo stanovit predispozici k HHT nebo HPAH u členů/jednotlivců v rizikové rodině.
Tento test je určen pro použití s lidskou DNA izolovanou z periferní krve, a ne pro použití s DNA izolovanou z čerstvého nádoru nebo tkáních fixovaných ve formalínu a zalitých v parafínu. Delece nebo duplikace detekované kitem P093 HHT/HPAH musí být ověřeny jinou metodou. Zejména delece nebo duplikace detekované pouze jedinou sondou vždy vyžadují ověření jiným způsobem.
Většina defektů v ENG, ACVRL1 and BMPR2 genech jsou bodové mutace a většina z nich nebude detekována metodou MLPA. Doporučuje se proto používat tento kit v kombinaci se sekvenační analýzou genů ENG, ACVRL1 a BMPR2. Tento test není určen k použití jako samostatný test pro klinické rozhodování. Výsledky tohoto testu musí být interpretovány molekulárním genetikem nebo ekvivalentním odborníkem.
Klinické pozadí
Hereditární hemoragická teleangiektázie (HHT)
Onemocnění s autozomální dominantní dědičností, které je charakterizováno přítomností více arteriovenózních malformací (AVM). U tohoto onemocnění se tepny propojují přímo s žílami, místo aby krev procházela přes kapiláry, což vede k vysokým hodnotám krevního tlaku. AVM se vyskytují na kůži, ale také v mozku, plicích, játrech a střevech. V závislosti na umístění, porušení těchto malformací může mít katastrofické následky pro pacienta. Diagnóza je stanovena na základě přítomnosti vícenásobných AVM v kůži, hlenových membránách nebo viscerálních orgánech.
Opakující se krvácení z nosu jsou také častým nálezem u pacientů s HHT. Molekulárně genetické testování se provádí k potvrzení nebo stanovení diagnózy u probandu. HHT je primárně způsobena patogenními změnami genu endoglin (ENG / HHT1) a aktivací A receptoru like typu 1 (ACVLR1 / HHT2). Oba geny kódují endoteliální buněčné povrchové receptory, které jsou součástí signální kaskády TGF-β / BMP, dráhy zapojené do angiogeneze, mezi několika dalšími vývojovými procesy. Až 10 % patogenních variant se skládá z velkých delecí/duplikací. Další informace naleznete na této adrese.
Hereditární plicní arteriální hypertenze (HPAH)
Dědičná autozomálně dominantní choroba. Toto onemocnění je způsobeno ztrátou nebo obstrukcí nejmenších plicních tepen, což vede k vysokému krevnímu tlaku v plicích. Diagnostika je založena na přítomnosti plicní hypertenze, která je potvrzena pomocí pravé katetrizace srdce a následnou identifikací heterozygotní patogenní varianty ve známém asociovaném genu (jednoduché případy) a / nebo potvrzením PAH u jednoho nebo více probandů v rodině.
Až 75 % HPAH je způsobeno variantou v genu pro kostní morfogenetický protein typu 2 (BMPR2). Z toho je 12-37 % způsobeno velkými duplikacemi/delecemi. Podobně jako geny ENG a ACVLR1, gen BMPR2 také kóduje buněčný povrchový receptor, který je součástí signální dráhy TGF-β / BMP. Sporadicky je PAH pozorován jako symptom HHT. Biologické podobnosti mezi kauzálními geny naznačuje podobnou etiologii mezi HPAH a HHT. To je podpořeno vzácnými pozorováními mutací v ACVLR1, a ještě častěji v ENG, způsobující HPAH.
V literatuře byla popsána pacientka s kombinovaným PAH a HHT fenotypem nesoucí deleci exonů 6 a 7 v BMPR2 (Handa a kol., 2014). Ve velmi vzácných případech HPAH mohou být způsobeny mutacemi v genu KCNK3, SMAD9 nebo CAV1. Podle našich znalostí, žádné HPAH kauzální delece nebo duplikace v těchto genech nebyly hlášeny. Více informací zjistíte zde.
Struktura genu
- Gen BMPR2 (13 exonů) pokrývá ~ 191 kb genomové DNA a nachází se na chromozomu 2q33.1-2, přibližně 203 Mb od p-teloméry.
- BMPR2 LRG_712 je totožný s GenbankNG_009363.1 a je k dispozici zde.
- Gen ACVRL1 (ALK1) (10 exonů) pokrývá ~ 16 kb genomové DNA a nachází se na chromozomu 12q13.3, přibližně 51 MB od p-teloméry.
- ACVRL1LRG_543 je totožný s Genbank NG_009549.1 a je k dispozici zde.
- Gen ENG (14 exonů) pokrývá ~ 40 kb genomové DNA a nachází se na chromozómu 9q34.11, přibližně 130 Mb od p-teloméry.
- ENG LRG_589 je totožný s Genbank NG_009551.1 a je k dispozici na této adrese.
Transkripční varianty
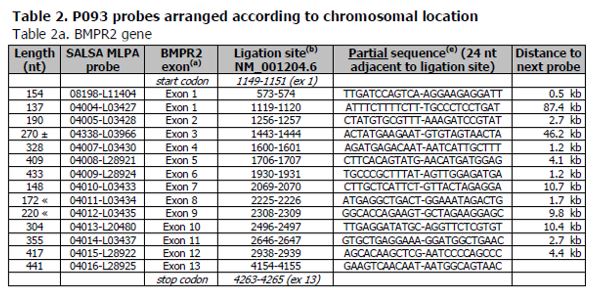
Jedna transkripční varianta BMPR2 byla popsána tady. BMPR2 transkripční varianty 1 (NM_001204.6; 12086nt; kódující sekvence 1149-4265) je referenčním standardem v projektu NCBI RefSeqGene. V tabulce 2 jsou ligační místa sond BMPR2 označena podle této sekvence, která obsahuje 13 exonů. ATG translační počátek je lokalizován v exonu 1 (1149-1151) a stop kodon je umístěn v exonu 13 (4263-4265).
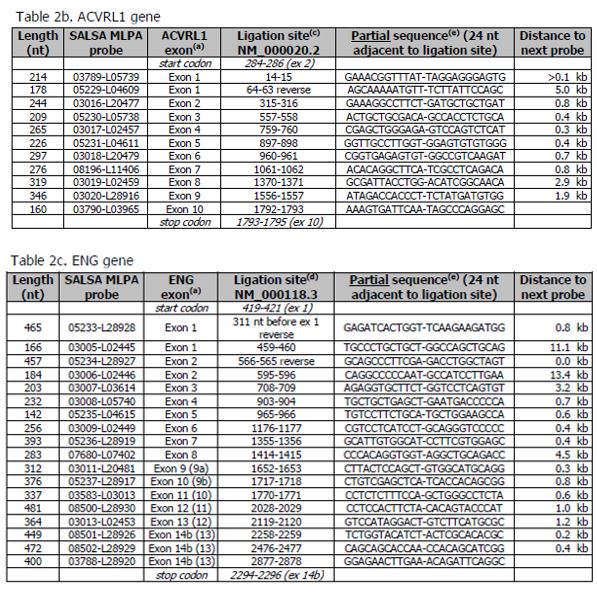
Byly popsány dvě transkripční varianty ACVLR1 tady. Sekvence ACVRL1 transkripční varianty 1 (NM_000020.2; 4263 nt; kódující sekvence 284-1795) je referenčním standardem v projektu NCBI RefSeqGene. V tabulce 2b jsou uvedeny místa ligace sond ACVRL1 podle této sekvence, která obsahuje 10 exonů. ATG translační počátek se nachází v exonu 1 (284-286) a stop kodon je umístěn v exonu 10 (1793-1795).
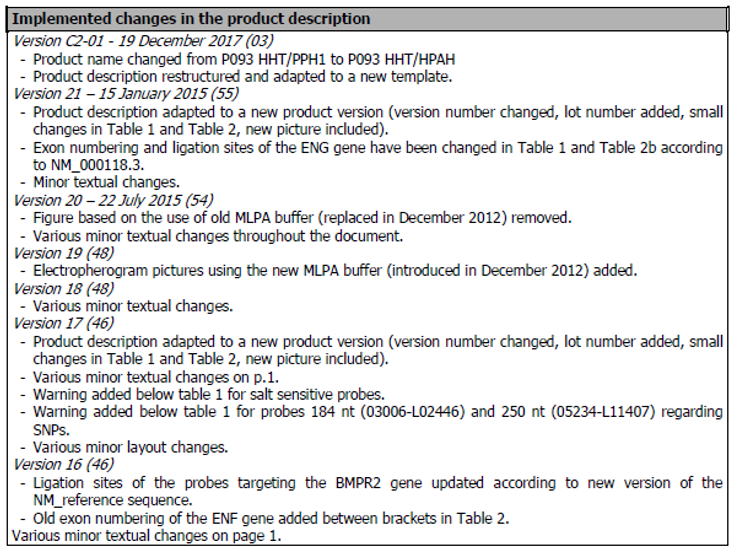
Byly popsány tři ENG transkripční varianty tady. Sekvence ENG transkripční varianty 2 (NM_000118.3; 3201 nt; kódující sekvence 419-2296) je referenčním standardem v projektu NCBI RefSeqGene. V tabulce 2c jsou uvedeny místa ligace sond ENG podle této sekvence, která obsahuje 14 exonů. ATG translační počátek je umístěn v exonu 1 (419-421) a stop kodon je umístěn v exonu 14(b) (2294-2296).
Číslování exonů
Číslování exonů použité pro geny BMPR2, ACVRL1, ENG v tomto popisu kitu P093-C2 HHT/PHAH a v analýze listu P093-C2 HHT/PHAH Coffalyser.Net je číslování exonů z RefSeq transcripts NM_001204.6, NM_000020.2 a NM_000118.3. Tyto sekvence jsou identické se LRG sekvencemi.
Obsah P093 HHT / HPAH kitu
Kit obsahuje 51 sond s amplifikačními produkty mezi 130 a 490 nt:
- 14 sond pro gen BMPR2 (jedna sonda pro každý exon a jedna další sonda pro exon 1),
- 11 sond pro gen ACVRL1 (jedna sonda pro každý exon a jedna další sonda pro exon 1),
- 18 sond pro ENG gen (jedna sonda pro každý exon a jedna další sonda pro exony 1 a 2 a dvě další sondy pro exon 14b),
- 8 referenčních sond.
Tento kit obsahuje devět kontrolních fragmentů, které generují amplifikační produkty mezi 64 a 105 nt:
- 4 DNA kvantitativní fragmenty (Q-fragmenty),
- 3 DNA denaturační fragmenty (D-fragmenty)
- 1 chromozóm X a jeden chromozóm Y-specifický fragment (Tabulka 1).
Q-fragmenty jsou viditelné pouze, pokud je použito méně než 100 ng DNA vzorku. Nízký signál 88 nebo 96 nt fragmentu naznačuje neúplnou DNA denaturaci. Více informací o tom, jak interpretovat výsledky z těchto kontrolních fragmentů, můžete nalézt v MLPA obecném protokolu.
Validace MLPA metody
Interní validace MLPA metody na 16 vzorcích DNA ze zdravých jedinců je nezbytná, a to zejména při prvním použití MLPA, nebo při změně zpracování vzorků, metodě extrakce DNA nebo používaného přístroje. Tato validace by měla vykazovat standardní odchylku <0,10 pro všechny sondy (s výjimkou 10 SNP-specifických sond) v experimentu.
Požadavky na vzorky
Izolovaná DNA z periferní krve bez nečistot, které ovlivňují MLPA reakci. Více informací naleznete v části o zpracování DNA vzorku v MLPA obecném protokolu.
Referenční vzorky
Referenční vzorky DNA by měly být odvozeny ze stejného typu tkáně, zpracovány za použití stejného postupu a připraveny za použití stejného způsobu, jako jsou DNA vzorky z pacienta. Referenční vzorky by měly být odvozeny od nepříbuzných jedinců, kteří jsou z rodin bez historie HHT nebo HPAH. Další informace týkající se výběru a použití referenčních vzorků lze nalézt v MLPA obecném protokolu.
Pozitivní kontrolní DNA vzorky
MRC-Holland nemůže poskytnout pozitivní vzorky DNA. Zahrnutí pozitivního vzorku v každém experimentu je doporučeno. Institut Coriell a DSMZ mají různorodou sbírku biologických zdrojů, které mohou být použity jako pozitivní kontrolní DNA vzorek ve vašich MLPA experimentech.
Identifikační čísla vzorků NA11213 (delece BMPR2), NA01229 (duplikace BMPR2) a NA10183 (duplikace ENG) od institutu Coriell byly testovány v MRC-Holland a mohou být použity jako pozitivní kontrolní vzorky. Kvalita buněčných linií se může změnit, proto vzorky by měly být před použitím validovány.
Charakteristika výkonu testu
Většina pacientů s HHT má patogenní varianty, které jsou snadno detekovány sekvenační analýzou. Odhaduje se, že přibližně 5 % všech pacientů s HHT má rozsáhlé přestavby genu ACVRL1 nebo ENG, zahrnující buď část genu nebo celý gen. Pokud se použije MLPA vedle sekvenační analýzy genů ACVRL1 a ENG, míra detekce se obecně zvyšuje o~ 10 %.
Většina pacientů s HPAH má patogenní varianty, které jsou snadno detekovány sekvenační analýzou. Odhaduje se, že přibližně ~ 12 % všech HHT pacientů má velké přestavby v genu BMPR2, zahrnující buď část genu nebo celý gen. Pokud se použije MLPA vedle sekvenační analýzy genu BMPR2, míra detekce se obecně zvyšuje o ~ 20 %.
Analytická citlivost a specifita (podle přehledu literatury 2005-2016) pro detekci delecí v BMPR2, ACVRL1 a ENG jsou velmi vysoké a lze je považovat za > 99 %.
Analytická výkonnost může být ohrožena:
- SNP nebo dalšími polymorfismy (např. indels) v cílové sekvenci DNA,
- nečistotami ve vzorku DNA,
- neúplnou DNA denaturací,
- použitím nedostatečného nebo příliš velkého množství vzorku DNA,
- použitím nedostatečných nebo nevhodných referenčních vzorků,
- problémy s kapilární elektroforézou nebo špatným postupem normalizace dat a dalšími technickými chybami.
MLPA obecný protokol obsahuje technické pokyny a informace o hodnocení/normalizaci dat.
Analýza dat
Coffalyser.Net software musí být použit pro analýzu dat v kombinaci s listem pro specifickou šarži - MLPA Coffalyser list. Nejnovější verze by měla být použita a je volně ke stažení na mlpa.com. Použití jiného ne-proprietárního softwaru může vést k neprůkazným nebo falešným výsledkům. Pro více informací o MLPA kontrole kvality a analýze dat viz Coffalyser.Net Manual.
Interpretace výsledků
Očekávané výsledky pro detekci BMPR2, ACVRL1 a ENG sondy detekující autozomální sekvence jsou počet kopií alel 2 (normální), 1 (heterozygotní delece) nebo 3 (heterozygotní duplikace). Standardní odchylka všech sond v referenčních vzorcích by měla být <0,10 a kvocient dávky (DQ) referenčních sond ve vzorcích pacientů by měl být mezi 0,80 a 1,20. Pokud jsou tato kritéria splněna, následující mezní hodnoty pro DQ sond (u těchto se očekává, že mají počet kopií 2) mohou být použity k interpretaci výsledků MLPA.
- Uspořádání sond podle chromozomální polohy usnadňuje interpretaci výsledků a může odhalit menší změny, jako jsou změny pozorované v případech mozaiky. Analýza rodičovských vzorků může být nezbytná pro správnou interpretaci komplexních výsledků.
- Falešně pozitivní výsledky: Vezměte prosím na vědomí, že abnormality zjištěné jedinou sondou (nebo více po sobě jdoucích sond) mají stále značnou šanci být falešně pozitivním výsledkem. Neúplná DNA denaturace (například z důvodu kontaminace solí) může vést ke snížení signálu sondy, zejména u sond umístěných v CpG ostrově nebo v blízkosti genu BMPR2. Použití dalšího kroku čištění nebo alternativní metody extrakce DNA může takové případy vyřešit.
- Falešné pozitivní výsledky - duplikace: Kontaminace DNA vzorků cDNA nebo PCR amplikony z jednotlivých exonů mohou vést k falešně pozitivním duplikacím (Varga et al., 2012). Analýza nezávisle odebraného vzorku sekundární DNA může vyloučit tento typ artefaktů.
- Normální počty kopií variant u zdravých jedinců jsou popsány v databázi genomových variant: http://dgv.tcag.ca/dgv/app/home. Uživatelé by měli vždy konzultovat nejnovější aktualizaci databáze a vědecké poznatky při interpretaci jejich výsledků.
Ne všechny odchylky zjištěné MLPA jsou patogenní. V některých genech, jsou známé intragenové delece a vedou k velmi mírné nebo žádné chorobě (Schwartz et al., 2007). Pro mnoho genů, existuje více než jedna transkripční varianta. Změny v počtu kopií exonů, které nejsou přítomny ve všech transkripčních variantách, nemusí mít klinický význam. Duplikace, které zahrnují první nebo poslední exon genu (například exony 1-3), mohou v některých případech mít za následek inaktivaci této kopie genu.
Změny v počtu kopií detekované referenčními sondami nebudou mít pravděpodobně žádný vztah k testovanému onemocnění.
Limitace metody
- Ve většině populací jsou hlavní příčinou genetických defektů v BMPR2, ACVRL1 a ENG genech malé (bodové) mutace, z nichž většina nebude detekována pomocí MLPA kitu P093 HHT/HPAH.
- MLPA nemůže zjistit žádné změny, které leží mimo cílovou sekvenci sond a nezjistí počet kopií neutrální inverze nebo translokace. I když MLPA nedetekuje žádné aberace, zůstává možnost, že existují biologické změny v tomto genu nebo chromozomální oblasti, ale zůstávají nedetekované.
- Změny sekvence (např. SNP, bodové mutace, malé indels) v cílové sekvenci sondy mohou způsobit falešně pozitivní výsledky. Mutace/SNP (i když> 20 nt od ligačního místa sondy) může snižovat signál sondy tím, že brání ligaci oligonukleotidové sondy nebo destabilizuje vazbu sondy na DNA.
Potvrzení výsledků
Změny v počtu kopií detekované pouze jedinou sondou vždy vyžadují ověření jinou metodou. Zřejmá delece detekovaná jedinou sondou může být způsobena např. mutací / polymorfismem, který zabraňuje ligaci nebo destabilizuje vazbu sondy na DNA. Sekvenační analýza může stanovit, zda mutace nebo polymorfismy jsou přítomny v cílové sekvenci sondy. Nalezení heterozygotní mutace nebo polymorfismu poukazuje na to, že dvě odlišné alely jsou přítomné ve vzorku DNA a falešně pozitivní výsledek MPLA lze získat.
Změny v počtu kopií detekované jednou nebo více než jednou po sobě jdoucích sond by měly být potvrzeny jinou nezávislou metodou, jako je long-range PCR, qPCR, array CGH nebo Southern blotting, kdykoli je to možné. Delece/duplikace delší než 50 kb mohou být často potvrzeny FISH.
Databáze mutací
Důrazně doporučujeme uživatelům ukládat pozitivní výsledky do těchto databází. Doporučení pro nomenklaturu popisující delece / duplikace jednoho nebo více exonů lze nalézt na této adrese. Prosím, oznamte změny v počtu kopií detekované referenčními sondami, falešně pozitivní výsledky kvůli SNP a neobvyklé výsledky (například duplikace BMPR2 exonů 6 a 8, ale ne exonu 7) do MRC-Holland: info@mlpa.com.
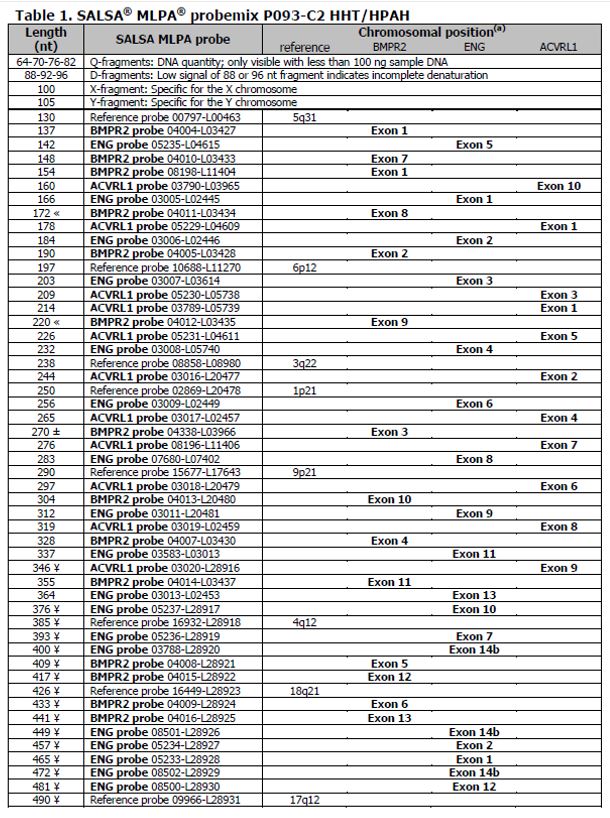
¥ Změněno ve verzi C2 (od šarže C2-1015 a dále). Změna délky, nebyla zjištěna žádná změna v sekvenci.
"Sonda umístěná uvnitř nebo blízko ostrova CpG. Nízký signál může být způsoben kontaminací soli v DNA vzorku vedoucí k neúplné denaturaci DNA, zejména v oblastech bohatých na CG."
± SNP rs863223425 by mohl ovlivnit signál sondy. V případě zjevných delecí se doporučuje sekvenování oblasti pro tuto sondu.
"Sonda umístěná uvnitř nebo blízko ostrova CpG. Nízký signál může být způsoben kontaminací soli v DNA vzorku vedoucí k neúplné denaturaci DNA, zejména v oblastech bohatých na CG.
(a)Číslování exonů pro geny BMPR2, ACVRL1 and ENG použité v tomto popisu kitu P093-C2 HHT/PHAH a v analýze listu P093-C2 HHT/PHAH Coffalyser.Net je číslování exonů z RefSeq transcripts NM_001204.6, NM_000020.2 a NM_000118.3. Sekvence jsou identické se LRG sekvencemi.
(b)Ligační místa pro BMPR2 gen v P093 HHT/HPAH MLPA jsou označeny podle Refseq sekvence NM_001204.6 obsahující 13 exonů.
(c)Ligační místa pro CVRL1 gen v P093 HHT/HPAH MLPA jsou označeny podle Refseq sekvence NM_000020.2 obsahující 10 exonů.
(d)Ligační místa pro ENG gen v P093 HHT/HPAH MLPA jsou označeny podle Refseq sekvence NM_000118.3 obsahující 14 exonů.
(e)Pouze část sekvence sond je uvedena. Kompletní sekvence sond jsou k dispozici na www.mlpa.com. Prosím, dejte nám vědět, pokud najdete nějaké chyby.
Zde použité číslování exonů se může lišit od literatury! Použili jsme číslování exonu ENG, které je uvedeno v NCBI NM_ referenčních sekvencích. V závorkách je číslování exonů, které je často používáno v literatuře. Kompletní sekvence sond jsou k dispozici na vyžádání: info@mlpa.com. Informujte nás prosím o chybách: info@mlpa.com.
Související SALSA MLPA kity
- P158 Juvenile Polyposis Syndrome: Obsahuje sondy pro SMAD4, BMPR1A a PTEN geny.